令和6年度 学際共創プロジェクト【バイオサイエンス部門・生命システム領域】
機械学習を用いたシングルセルRNA-seqデータからの高精度タンパク質発現量予測
研究代表者: 奥崎 大介(免疫学フロンティア研究センター)
研究分担者: 瀬尾 茂人(情報科学研究科/IDS)、石川 昌和(香川大学)
研究の背景
シングルセル RNA シーケンシング(scRNA-seq)は細胞ごとの転写物(RNA)を網羅的に取得できる一方で、捕捉できるRNA分子数が限られるため、発現していても検出されない遺伝子が多いという課題があります。また、同一遺伝子から生じるタンパク質アイソフォームを配列情報だけで識別することも難しく、この結果、細胞機能や状態の差異を見落とす恐れがあります。
こうした弱点を補うために、RNAと細胞表面タンパク質を同時に定量するCITE-seqが開発されました。タンパク質は翻訳後修飾やアイソフォームの違いを反映した最終的な機能分子であるため、CITE-seqを使用すれば、scRNA-seqでは区別できなかった表面タンパク質アイソフォームを直接測定でき、細胞状態をより正確に捉えることができます。しかし、CITE-seqは測定できるタンパク質が抗体パネルに依存し、また試薬コストが高いため、Human Cell Atlasのような大規模プロジェクトでも依然としてRNA単独計測が主流です。
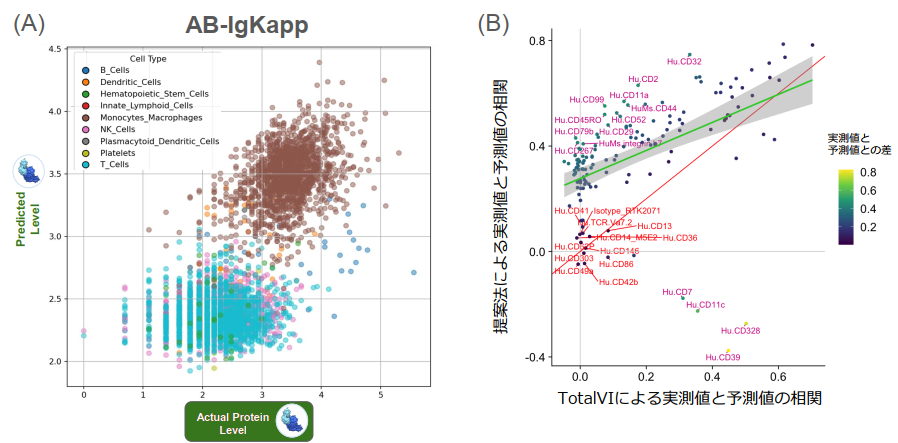
研究の目的
CITE-seqデータは「RNAとタンパク質の発現量の対応関係」が対になった教師データとして利用できる点で極めて価値が高いです。このペア情報を使用することで、RNA発現プロファイルからタンパク質発現量を高精度に予測するモデルを構築できれば、CITE-seqを実施していないscRNA-seqデータにも擬似的にタンパク質情報を付加することが可能になります。
これにより、抗体パネルが異なる複数データセットの統合解析、タンパク質依存的な細胞型同定やシグナル経路推定、さらには治療標的候補のスクリーニングなど、幅広い応用が期待されます。そのため、RNAだけからタンパク質量を推定する計算手法の開発は、実験コストを抑えつつ細胞機能を深く理解するための鍵となります。
本研究では、CITE-seq由来のデータを訓練に利用し、RNA情報のみから細胞表面タンパク質量を高精度に推定する計算モデルを構築することを目的とします。
本年度の成果について、詳しくは活動報告書(PDF)をご覧ください。
